Helicobacter pylori è un importante patogeno che infetta l’uomo ed è stato riconosciuto dalla Organizzazione Mondiale della Sanità come cancerogeno di classe I.
La capacità di questo batterio Gram-negativo di colonizzare l’ostile nicchia dello stomaco e di stabilire un’infezione persistente dipende dall'espressione coordinata di fattori di virulenza che consentono al patogeno di adattarsi alle avverse condizioni ambientali e di contrastare i meccanismi di difesa dell’ospite.
La ricerca svolta dal nostro gruppo si avvale di metodologie di biologia molecolare, biochimiche, e genetiche per studiare i meccanismi molecolari adottati da due repressori trascrizionali coinvolti nella risposta heat-shock, HrcA e HspR, di un regolatore di risposta essenziale per la vitalità del batterio chiamato HP1043, coinvolto nella regolazione di cruciali processi cellulari, e di un piccolo RNA di regolazione coinvolto nella motilità batterica, chiamato CncR1.
Temi di ricerca del gruppo
Controllo Trascrizionale in Risposta a Heat-shock:
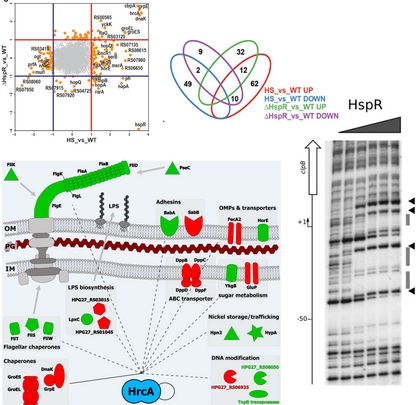
La risposta heat-shock nel patogeno umano H. pylori è governata da due repressori trascrizionali, HrcA e HspR. Questi regolatori, in condizioni di crescita normali mantengono la repressione dei principali geni heat-shock e in seguito a shock termico permettono un rapido e transitorio accumulo dei trascritti regolati. Il nostro gruppo studia i meccanismi molecolari esercitati da ciascun regolatore e le loro interazioni per controllare la trascrizione dei geni in risposta a fluttuazioni di temperatura.
Controllo Trascrizionale del Regolatore Essenziale HP1043:
Il regolatore trascrizionale HP1043 è codificato da un gene essenziale per la vitalità del batterio. L’impossibilità di generare un mutante di delezione per il gene hp1043 o di modulare i livelli proteici del regolatore in vivo ha fino ad ora impedito la comprensione della sua funzione. Attraverso saggi di immunoprecipitazione della cromatina (ChIP) abbiamo identificato a livello genomico 37 promotori legati in vivo da HP1043 e determinato in vitro le sequenze consenso di legame. Studiamo i meccanismi molecolari di attivazione della trascrizione in vitro identificando i determinanti di specificità del legame al DNA e gli effetti sulla sua capacità di attivare la trascrizione. In parallelo, costruiremo un mutante condizionale di hp1043 che ci permetterà di definire l’impatto del silenziamento dell’espressione del regolatore sull'intero trascrittoma batterico.
Inoltre, dato il ruolo cruciale di HP1043 che lo rende un target antibatterico molto promettente, stiamo conducendo studi di docking proteina-DNA e di dinamica molecolare finalizzati allo screening virtuale di librerie di molecole per l’identificazione di composti capaci di legare e interferire con l’attività della proteina HP1043 e di conseguenza con la vitalità di H. pylori.
Definizione del ruolo regolativo e del meccanismo molecolare di RNA non-codificanti in H. pylori
Recenti studi hanno dimostrato che H. pylori esprime un numero molto alto di trascritti non codificanti che potrebbero svolgere un ruolo regolativo attraverso svariati meccanismi. Nel nostro gruppo ci stiamo occupando dello studio di alcuni di questi RNA, combinando metodologie molecolari (generazione di mutanti, saggi di interazione RNA-RNA, structure probing) e approcci di genomica funzionale (RNA-seq, studi di proteomica comparativa e altri). Ad esempio, per definire il ruolo regolativo di CncR1, un RNA non-codificante regolato da HP1043, abbiamo implementato un approccio di genomica funzionale recentemente sviluppato chiamato MAPS (purificazione dell'affinità MS2 accoppiata con il sequenziamento dell'RNA), per definire l'interattoma di CncR1.