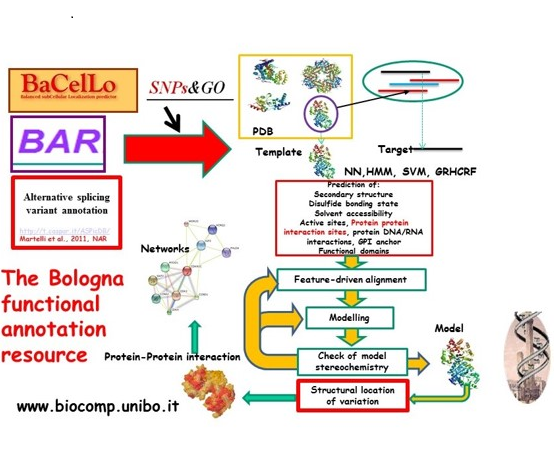
Temi di ricerca del gruppo
Sviluppo di algoritmi per l’annotazione strutturale e funzionale di proteine e loro varianti. Il problema principale dell’ “era genomica” è la continua produzione di dati in diversi domini della complessità cellulare. È fondamentale estrarre dai dati l’informazione utile per la comprensione delle regole di organizzazione gerarchica della complessità biologica che giustifichino l’emergenza delle proprietà fisiologiche a partire dalle singole molecole.
L’informazione non ridondante estratta da banche dati è analizzata con algoritmi di tipo statistico, probabilistico o di apprendimento automatico al fine di inferire proprietà strutturali e funzionali per molecole poco studiate sperimentalmente. Il risultato dell’indagine è un algoritmo, noto in gergo come predittore, che può fornire la probabilità che una determinata proprietà sia o non sia associabile alla molecola in esame (http://www.biocomp.unibo.it/predictors.html)
Interazione tra proteine in organismi diversi e loro integrazione in modelli di biologia di sistema. La chiave di lettura della complessità biologica è l’interazione strutturale e funzionale tra molecole nei compartimenti cellulari.
L’interazione tra proteine è alla base dei processi metabolici e biologici. La modificazione di alcune delle componenti a partire dal genoma può causare perdita di funzionalità a livello sistemico. Tali analisi sono supportate da tecniche di enrichment che identifica le funzioni statisticamente significative associate a un gruppo di geni o varianti (http://net-ge.biocomp.unibo.it/)
Relazione genotipo-fenotipo. Integrazione di dati strutturali, genomici e funzionali per la relazione tra genotipo e fenotipo.
È possibile formulare ipotesi sulla relazione tra genotipo e fenotipo per l’uomo e per le sue malattie calcolando quali annotazioni strutturali e funzionali siano condivise, in maniera statisticamente significativa, tra proteine associate allo stesso fenotipo e evidenziando il loro comune coinvolgimento in processi biologici e vie metaboliche. Questa analisi permette di fornire una visione integrata della complessità delle relazioni tra genotipo e fenotipo (http://edgar.biocomp.unibo.it/gene_disease_db/ ; http://edgar.biocomp.unibo.it/phenpath )
Sviluppo di modelli molecolari per fenotipi complessi. L’insorgere di una malattia può dipendere da un insieme di fattori a livello molecolare, spesso individuo-dipendenti.
L’integrazione delle mutazioni ereditarie e somatiche associate a varie patologie, in particolare quelle tumorali, permette l’individuazione di possibili meccanismi molecolari che possono suggerire ulteriori approfondimenti sperimentali. I dati sono di solito ottenuti da esperimenti di Next generation sequencing (NGS) e resi disponibili dai consorzi pubblici. L’analisi dei dati in un’ottica di genomica funzionale permette lo sviluppo di modelli che forniscono la probabilità di rischio relativa all’insorgenza del fenotipo in funzione delle mutazioni osservate.
Aspetti della relazione struttura-funzione in ATPasi di membrana. Relazione tra il numero di protoni per molecola di ATP traslocati attraverso la membrana e il numero di subunità transmembrana nelle ATPasi di tipo F.
Le ATPasi di tipo F sono assimilabili a motori nanoscopici rotazionali, nei quali un rotore transmembrana viene mosso da un gradiente protonico relativamente a uno statore extramembrana che sintetizza ATP. Abbiamo messo a punto un metodo per determinare sperimentalmente il numero di protoni vengono traslocati per molecola di ATP sintetizzata. Recentemente abbiamo ottenuto evidenze che tale parametro è variabile e dipende dallo stato energetico della cellula.